本文通過實際案例,展示了如何使用實體表格和應用模板,來批量投遞的10個樣本的分析運行任務。
假設用戶有10個全基因組測序的樣本數據需要分析,我們通過以下步驟詳細介紹如何通過基因分析平臺來“一鍵”高效簡便的完成所有工作。
創建工作空間,獲取分析應用
創建batch-submit-demo工作空間,如已有可用工作空間也可忽略該步驟。
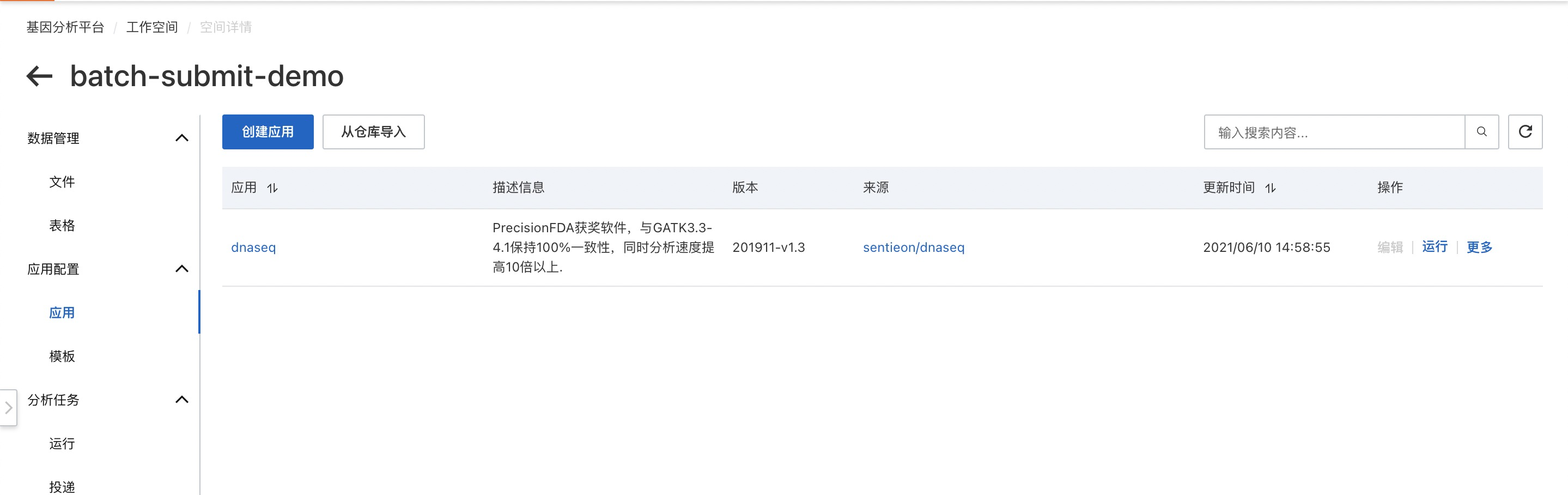
在應用倉庫中安裝sentioen/dnaseq到該工作空間中。安裝成功后,跳轉到對應工作空間的應用列表。
上傳基因數據文件
使用命令行工具ossutil64,將本地文件上傳到工作空間對應的OSS Bucket中。
root@demo:~/batch-submit-demo# tree reads/
reads/
├── S001_R1.fastq.gz
├── S001_R2.fastq.gz
├── S002_R1.fastq.gz
├── S002_R2.fastq.gz
├── S003_R1.fastq.gz
├── S003_R2.fastq.gz
├── S004_R1.fastq.gz
├── S004_R2.fastq.gz
├── S005_R1.fastq.gz
├── S005_R2.fastq.gz
├── S006_R1.fastq.gz
├── S006_R2.fastq.gz
├── S007_R1.fastq.gz
├── S007_R2.fastq.gz
├── S008_R1.fastq.gz
├── S008_R2.fastq.gz
├── S009_R1.fastq.gz
├── S009_R2.fastq.gz
├── S010_R1.fastq.gz
└── S010_R2.fastq.gz使用ossutil64將本地reads文件上傳到基因分析平臺中。
root@demo:~/batch-submit-demo#
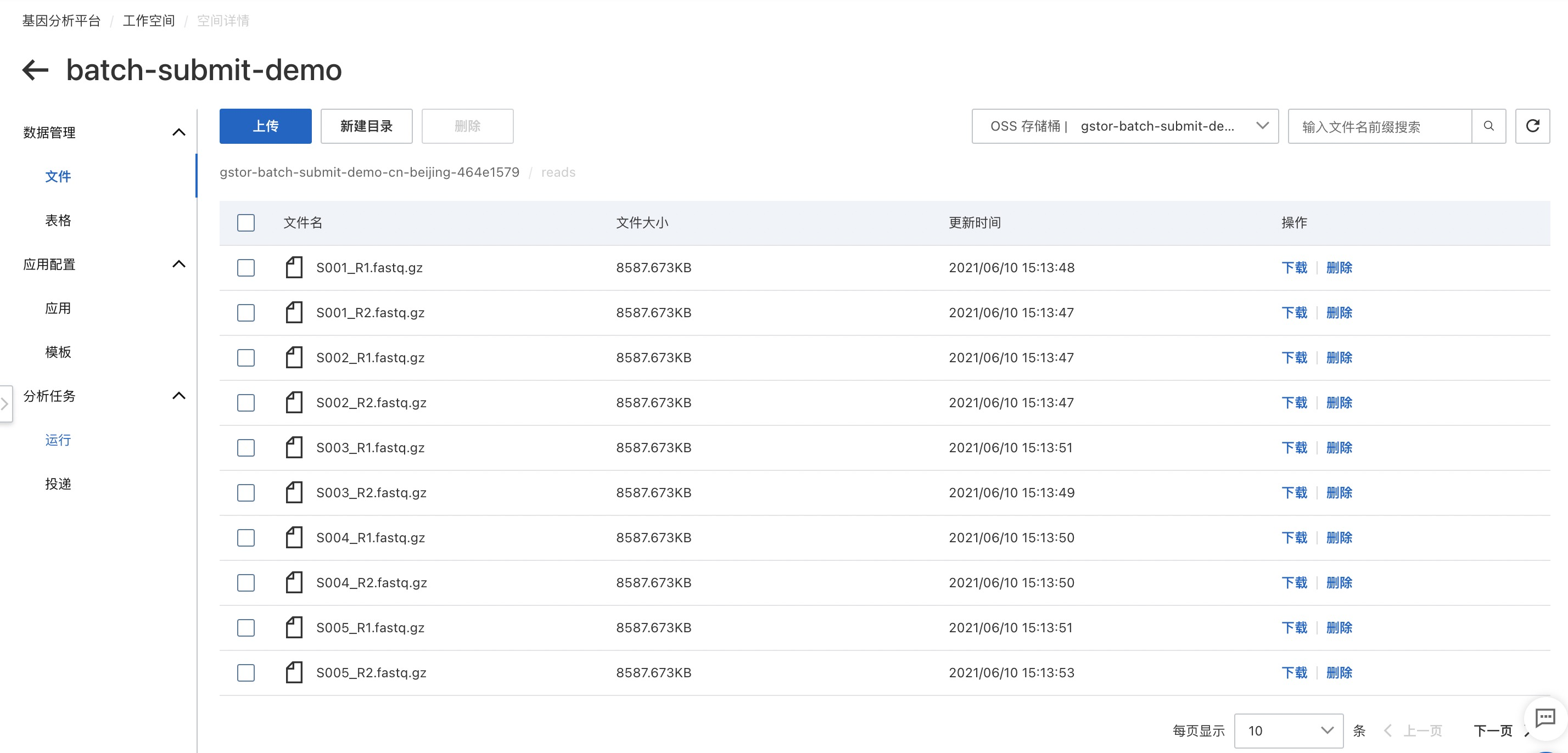
~/ossutil64 cp -e oss-cn-beijing.aliyuncs.com -r reads/ oss://gstor-batch-submit-demo-cn-beijing-464e1579/reads/上傳完成后,在工作空間中可以瀏覽文件。
定義實體表格,并添加數據
按照演示的目的,我們定義了dnaseq分析應用輸入所需要的樣本屬性。
name:應用的名字
reads_group: 測序數據所屬的reads group
FASTQ1:樣本測序數據的read_1
FASTQ2: 樣本測序數據的read_2
在“工作空間 -表格”中添加sample的實體類型。
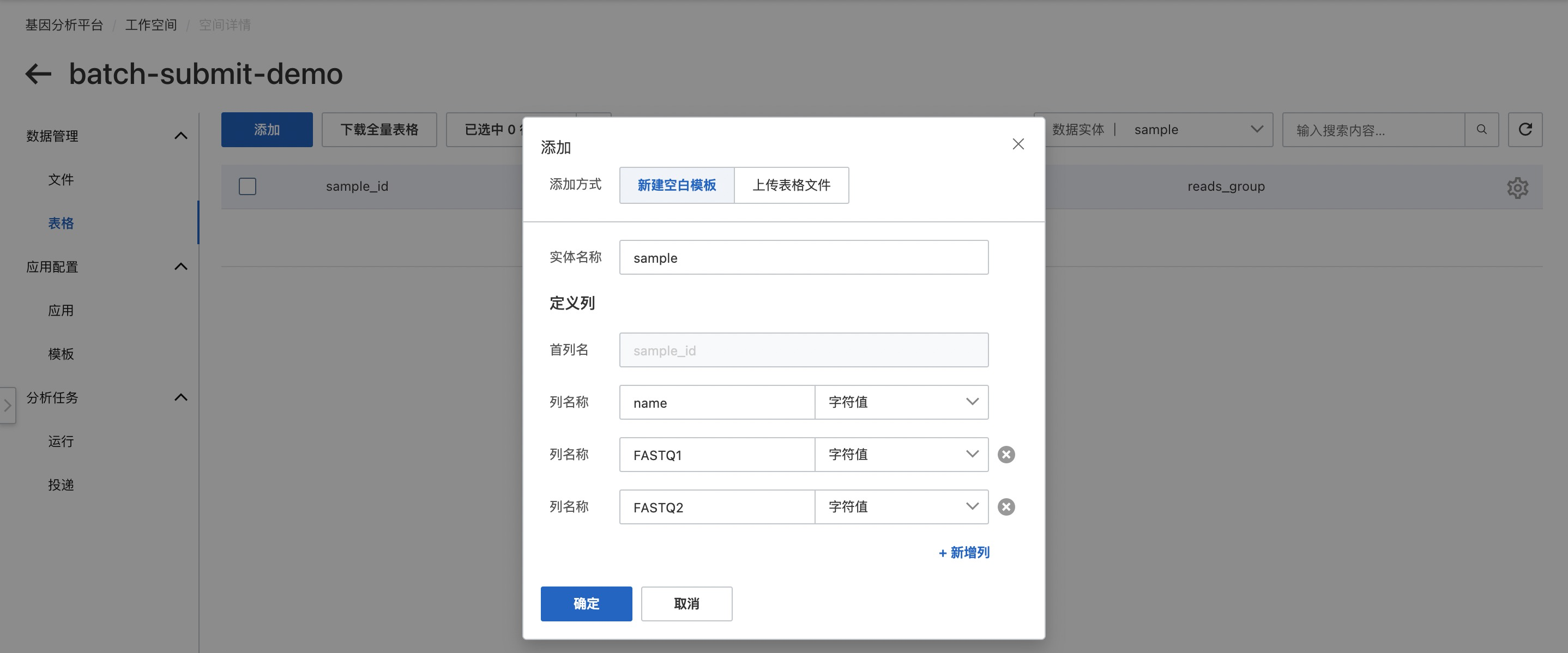
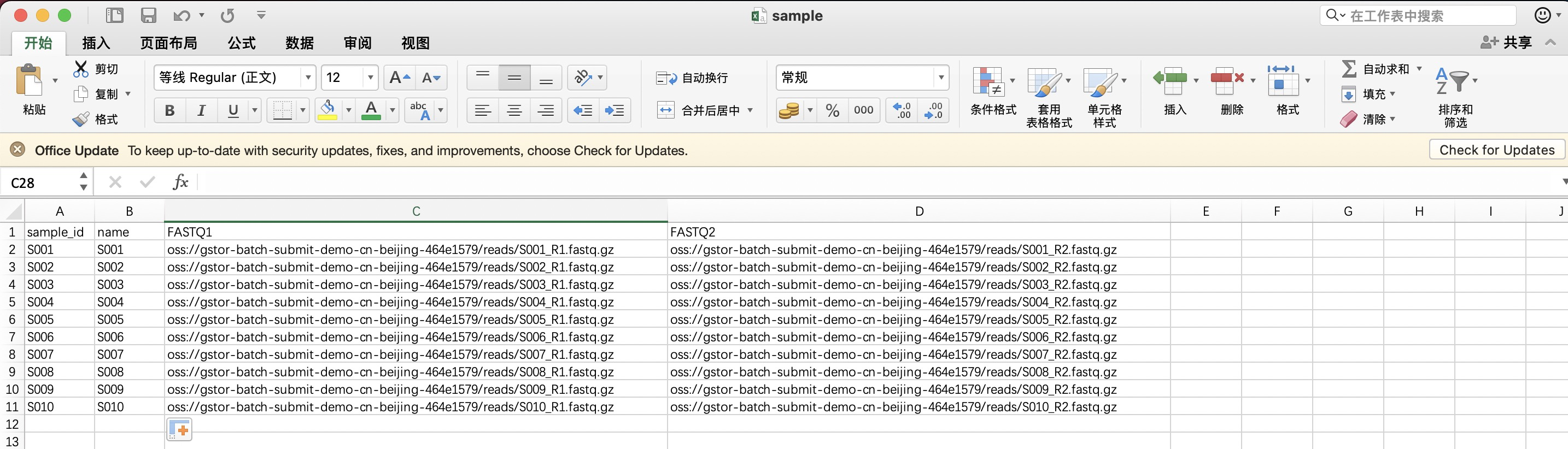
下載“sample.csv”模板后,在本地使用Excel進行編輯,將每個樣本實際對應的信息和測序文件OSS地址編輯保存。
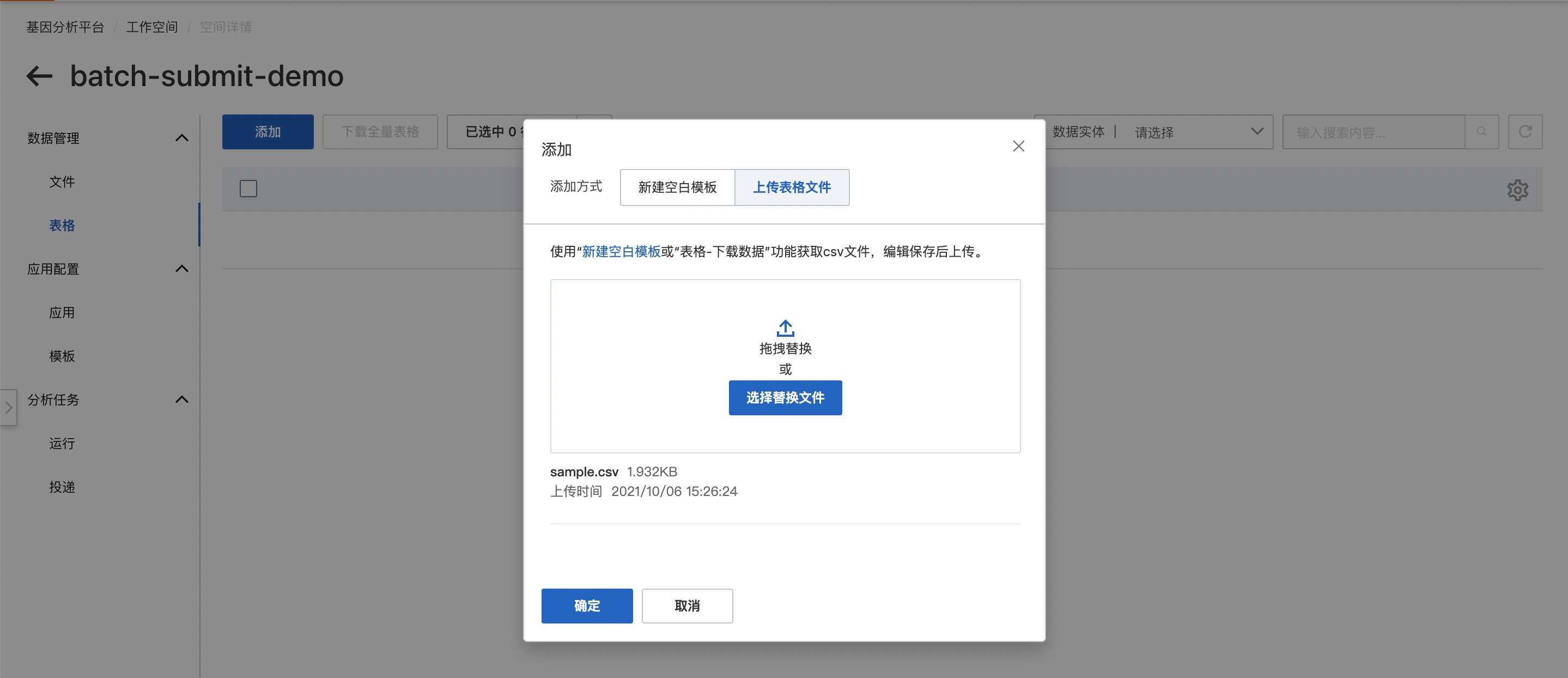
上傳sample.csv文件到空間工作的表格中。
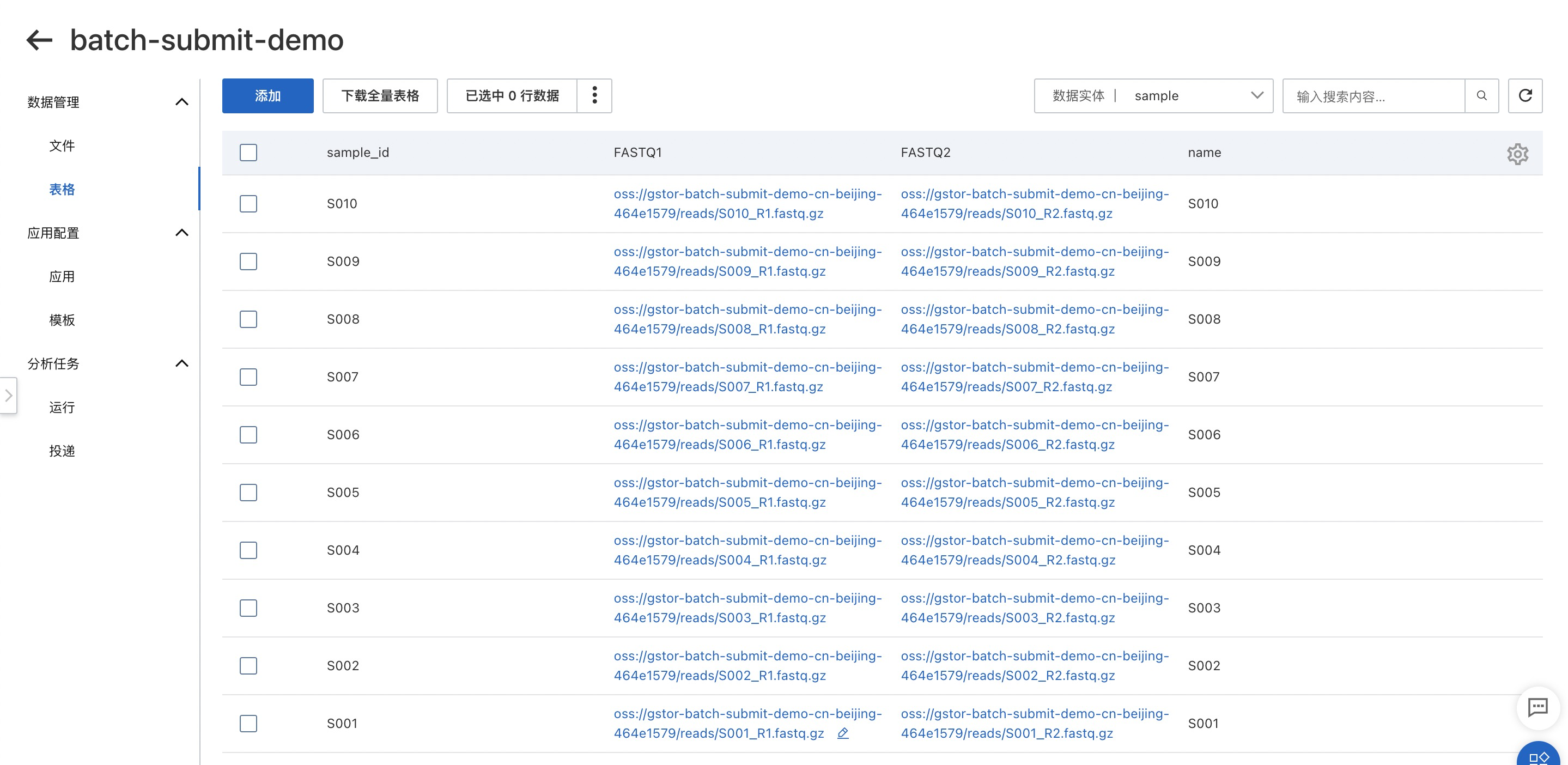
查看sample實體表格數據。
創建應用運行模板
在應用頁面中選擇dnaseq應用來創建分析模板。點擊右側“更多”,選擇創建模板:

選擇sample作為模板的根實體類型。
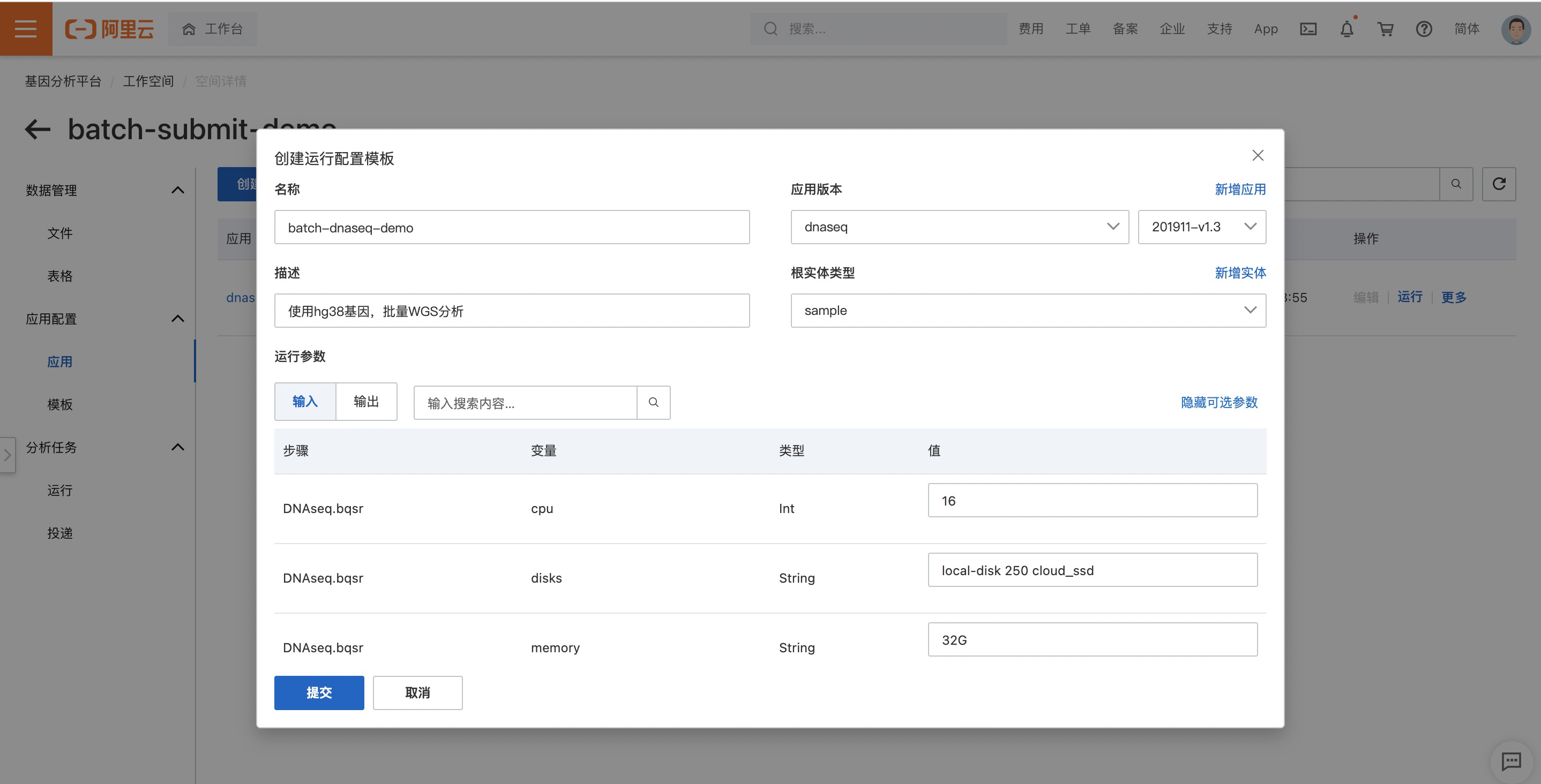
對于dnaseq應用分析的必須參數,通過實體表達式和固定值來填充,確保滿足運行條件。
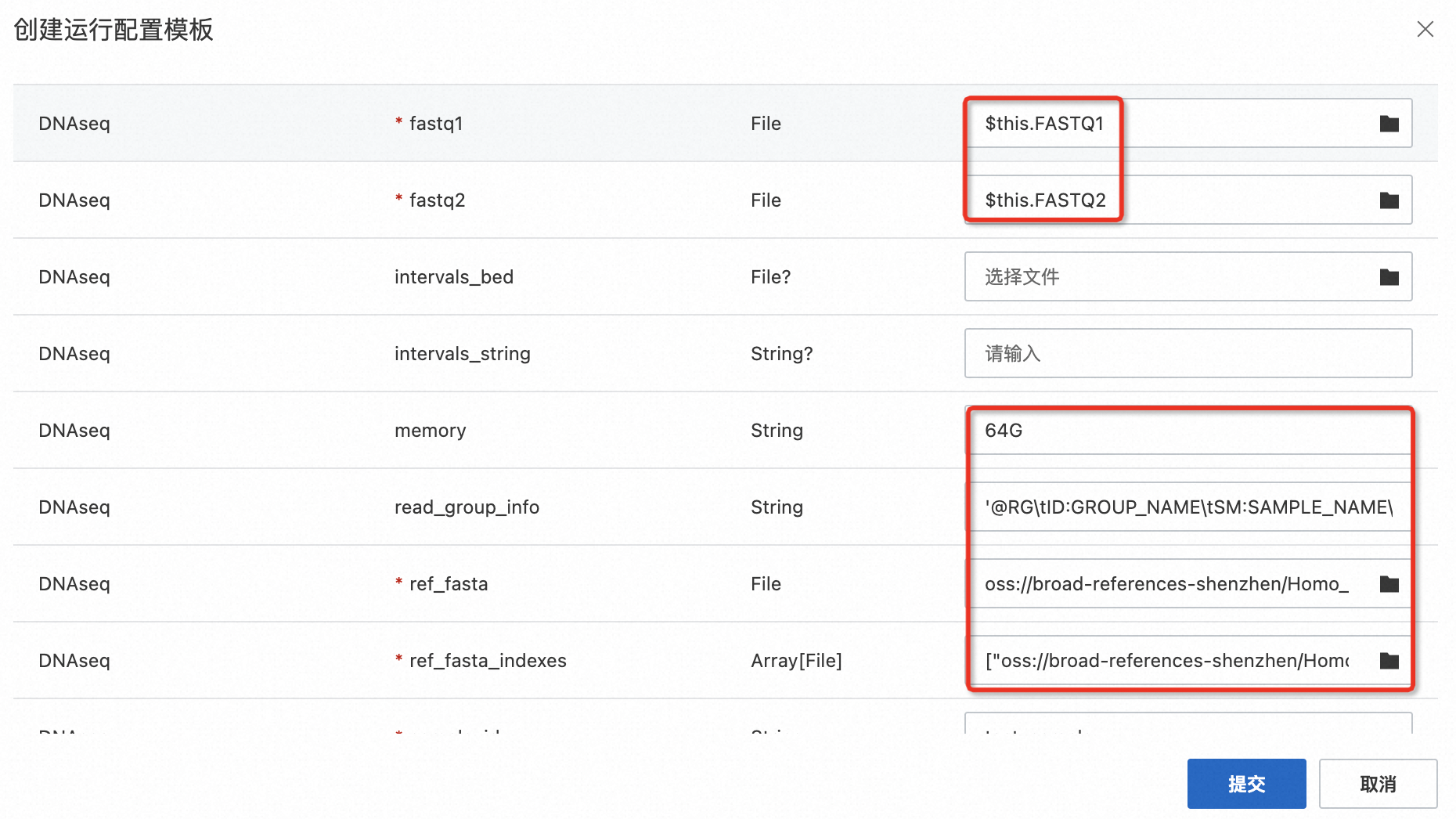
同時可以直接在輸出中定義新的表格列,來保存運行任務完成后的分析結果。
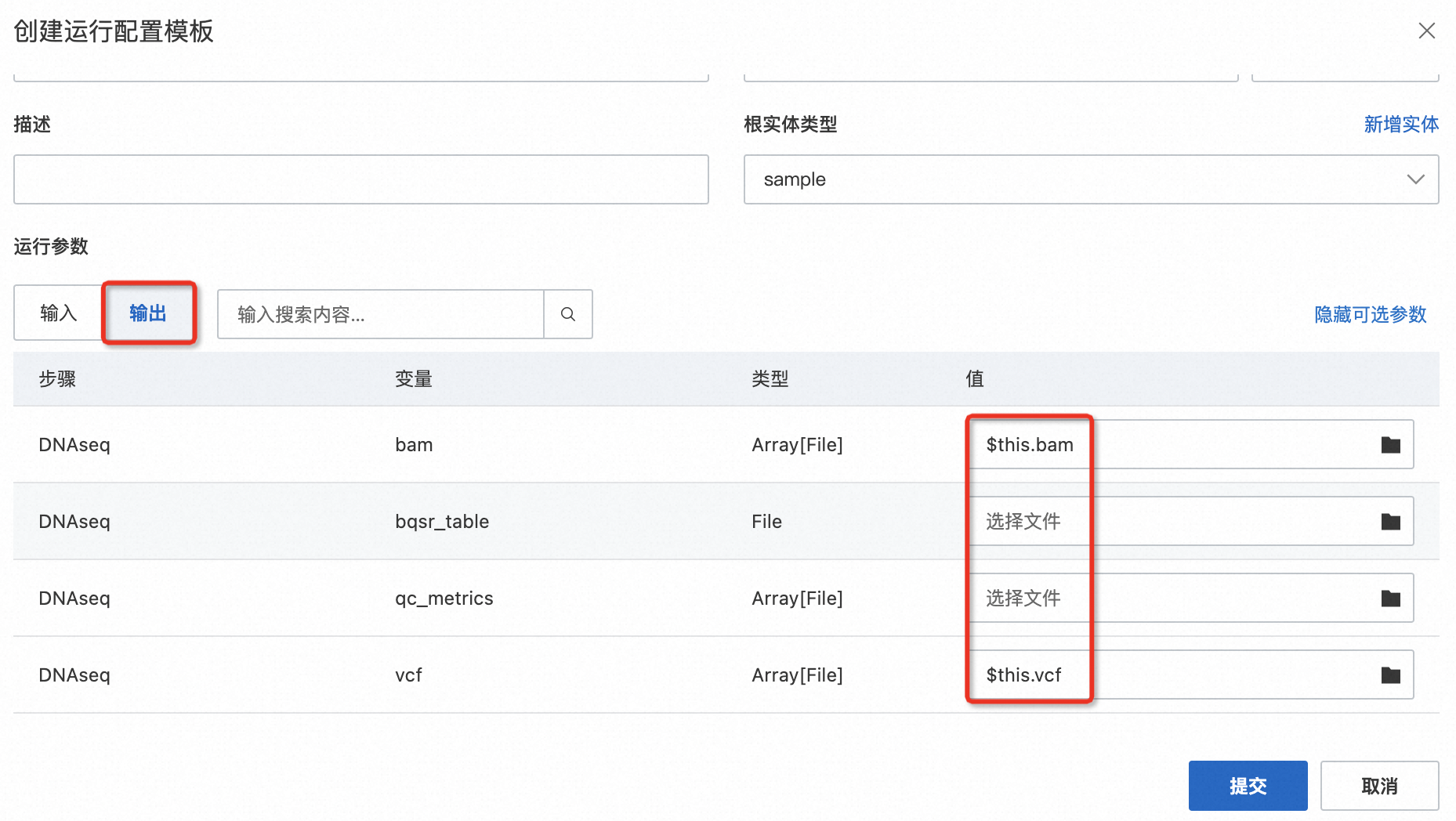
提交后即可獲得為指定應用設計好的運行模板。
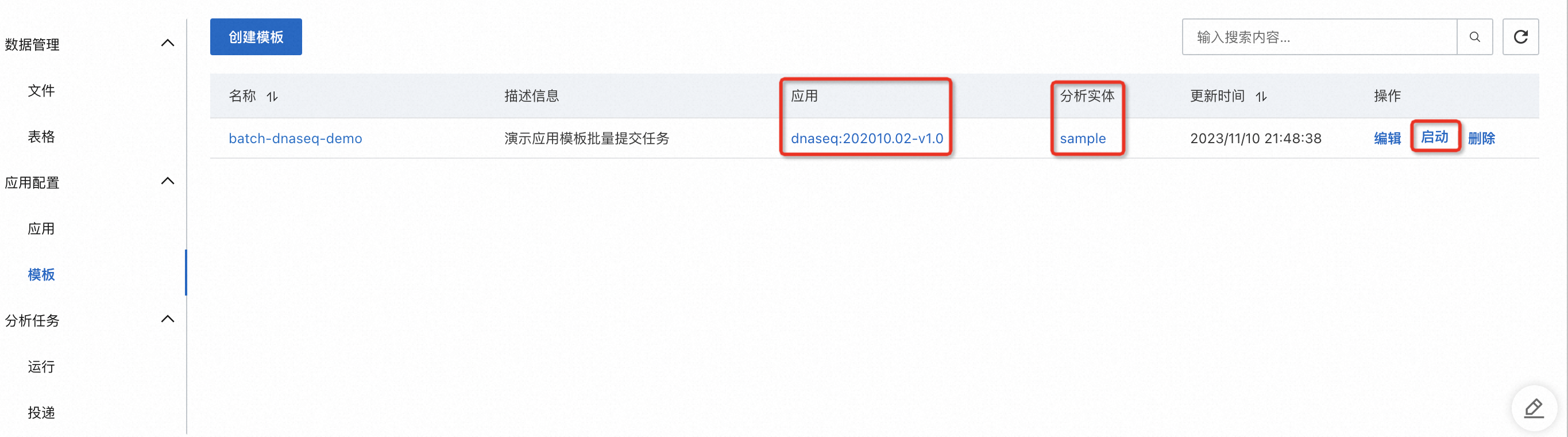
批量投遞10個樣本的分析運行任務
點擊“啟動”模板,只需要選擇需要分析的樣本(sample)即可投遞任務,無需再指定其他運行參數。
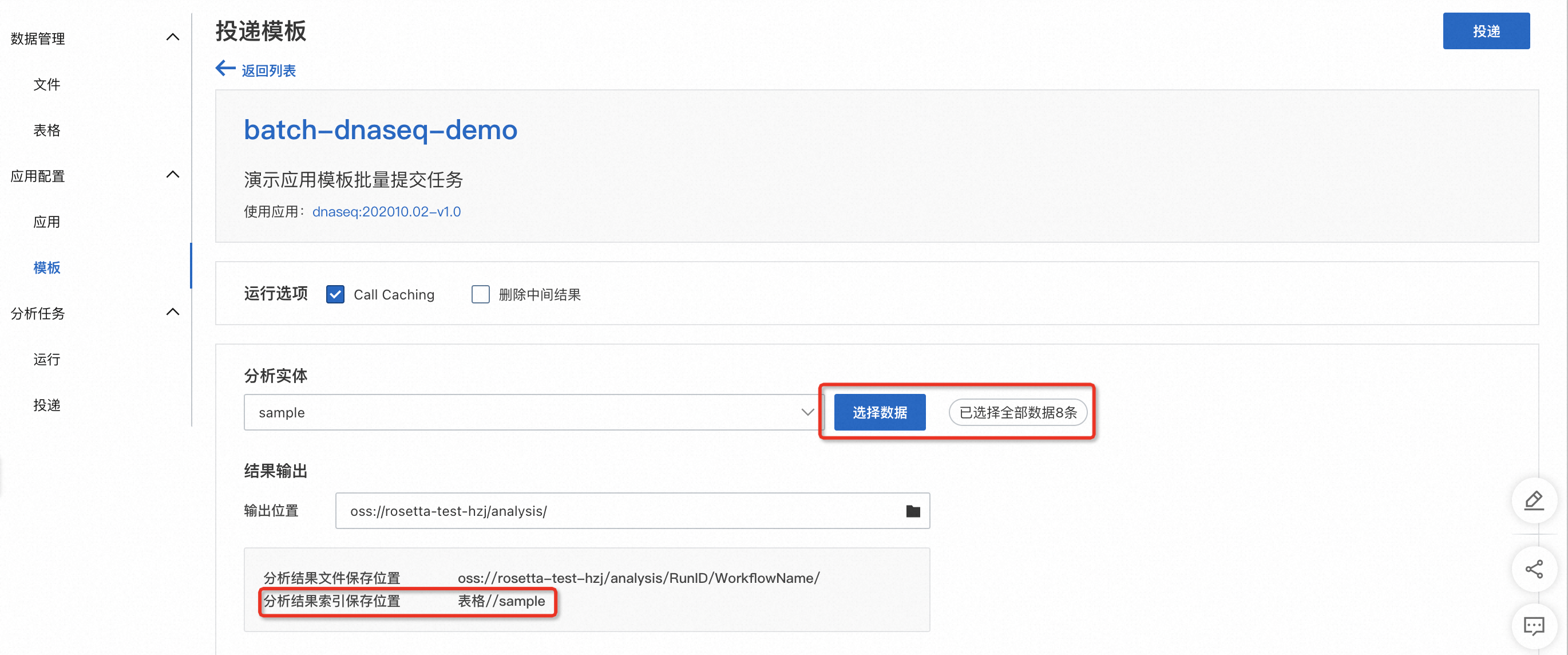
通過投遞ID管理所有運行任務
投遞完成后,即可生產投遞記錄。通過暫定和恢復投遞記錄,即可批量管理該投遞解析生成的所有運行任務。
完成基因數據分析任務,查看結果
運行任務的結果會被寫回表格中,可以通過查看或下載表格,獲得這一批樣本的分析結果。